
Système nerveux du derme segmenté (projection)
Texte présenté à la 28ème Journée ISS France (ENSMP, 3 Février 2005) (avec quelques modifications)
Gabriel Fricout, Dominique Jeulin (ENSMP
CMM),
John Bertram, Luise McCullen, Richard
Young (Dept of Anatomy and Cell Biology, MU),
Gahnim Almahbobi (Monash Institute),
Michael Cameron (Monash University)
Christophe
Gratin (ADCIS), Thibauld Nion (ENSMP CMM)
ENSMP CMM - 35, rue
Saint-Honoré 77300 Fontainebleau, France
Monash University (MU) - Clayton,
Victoria, Australia, 3800
ADCIS - 10 Avenue
de Garbsen, 14200 Hérouville-St-Clair, France
Des algorithmes de squelettisation ont été développés pour étudier la structure des arbres urinaires de souris de façon automatique. Ils ont été conçus grâce à la collaboration du département d'Anatomie et de Biologie Cellulaire de Monash University (Melbourne, Australie) avec le Centre de Morphologie Mathématique de l'École des Mines de Paris (Fontainebleau, France), et publiés au début 2000.
Ces algorithmes ont, depuis, été testés puis utilisés pour diverses études sur le rein (équipe de J. Bertram), puis sur la prostate (équipe du Monash Institute). Il apparait aujourd'hui que ces algorithmes sont assez robustes et capables de s'adapter à des problèmes variés mettant en jeux l'analyse d'image de structures arborescentes en trois dimensions.
Cette présentation rappèle le fonctionnement général de ces algorithmes, et donne une illustration consacrée à la squelettisation du système nerveux du derme de rats (image fournie par Santos Carvajal-Gonzalez) qui donnera lieu à leur intégration au logiciel Aphelion™.

Système nerveux du derme segmenté (projection)
Les étapes de fonctionnement de ces algorithmes, appliqués à une image segmentée, sont les suivantes:
Les extrémités de l’arbre détectées sont les maxima locaux de la fonction distance géodésique calculée à partir d'un point quelconque à l’intérieur de l’objet. Pour être plus robuste, cette fonction est pondérée par une fonction inversement proportionnelle à la “distance à l'extérieur”, dans le cas du rein et du derme.
Les extrémités sont reliées une à une au squelette en construction, en créant une branche qui emprunte le plus court chemin géodésique entre le squelette et l'extrémité. Cette distance est pondérée par la fonction “distance à l'extérieur” pour éviter que le squelette longe les bords de l’objet.
Le résultat est un objet mince centré dans la structure et sur lequel il est facile de déterminer les points de branchement, et de calculer les longueurs des branches. La récolte de toutes ces données permet de ensuite de réaliser des études quantitatives sur un grand nombre de spécimen et d'en tirer par exemple des résultats statistiques et des modèles d’arbres aléatoires.
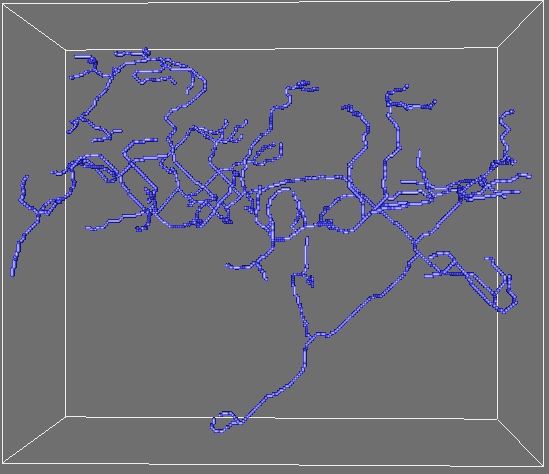
Squelette 3D de l'image du derme (obtenu par le logiciel Aphelion(TM)